发现新的烟酰胺腺嘌呤-RNA去帽酶


日期:2020-01-02
来源:BioArt 浏览:
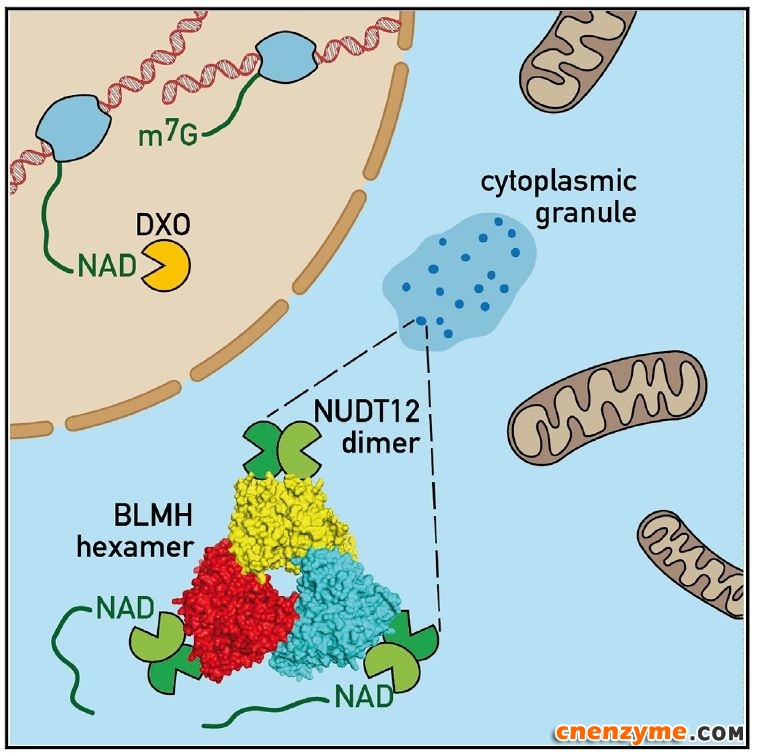
1211 评论:0 核心提示:2019年12月24日,日内瓦大学分子生物学系Ramesh Pillai组的乌皓、李灵运、陈冠鸣等研究人员在Cell Reports杂志上发表了题为Decapping Enzyme NUDT12 Partners with BLMH for Cytoplasmic Surveillance of NAD-Capped RNAs 的研究成果。发现了新的NAD-RNA的去帽酶NUDT12。与DXO不同的是,NUDT12通过水解烟酰胺腺嘌呤二磷酸的磷酸键,从而移除烟酰胺帽子结构。
在真核生物中,RNA聚合酶II参与转录的RNA分子在其转录过程中,会在5’端进行带帽修饰,通过三磷酸键连接一个7位甲基化的鸟嘌呤(m7G)。这种m7G 帽子结构早在40多年前就被科学家们解析,并发现其在RNA剪接、出核及维持RNA稳定等各个环节都发挥着重要的作用【1】。长期以来,这种m7G帽子结构被认为是真核生物唯一的带帽形式。然而,随着检测技术灵敏度的提高,在最近的十年中,其它类型的RNA“帽子”结构开始被科学家们发现。在众多非m7G帽子结构中,首先被人们发现的是烟酰胺腺嘌呤 (NAD)帽子结构。它在2009年首次从大肠杆菌RNA中发现【2】,后来陆续在酵母、细胞系、植物和小鼠组织中被检测到【3-7】。与m7G帽子结构的转录后修饰不同的是,NAD帽子是随同RNA转录同时进行的【7】。在转录起始的时候,用NAD代替了经典的核糖核酸。NAD-RNA在细胞中的含量极其微少,仅为m7G-RNA的千分之一左右,且能够发生RNA剪接和多聚腺苷化等RNA合成的事件【5,6】。然而NAD-RNA的分布和功能至今仍然未知。
在RNA的降解过程中,RNA去帽是其必备的过程。在m7G-RNA的降解过程中,DCP2是其去帽酶【8】。DCP2通过水解m7G三磷酸中的磷酸键,解离5’端单磷酸RNA。这种解除帽子结构的单磷酸RNA随后会被核酸外切酶XRN1水解。对于NAD-RNA的去帽酶,人们的认知还较少。在2017年,有研究小组发现在真核生物的细胞核内,DXO可以通过水解整个NAD来完成NAD-RNA的脱帽5。但研究同时指出,并非所有NAD-RNA都可以通过这一方式脱帽。且由于NAD-RNA存在出核的可能性,其在细胞质内的监管机制尚不为人所知。
2019年12月24日,日内瓦大学分子生物学系Ramesh Pillai组的乌皓、李灵运、陈冠鸣等研究人员在Cell Reports杂志上发表了题为Decapping Enzyme NUDT12 Partners with BLMH for Cytoplasmic Surveillance of NAD-Capped RNAs 的研究成果。发现了新的NAD-RNA的去帽酶NUDT12。与DXO不同的是,NUDT12通过水解烟酰胺腺嘌呤二磷酸的磷酸键,从而移除烟酰胺帽子结构。
研究发现,NUDT12定位于细胞质中的一种未知的granule结构中,并且是以同源二聚体的形式存在。这种同源二聚作用对于其稳定性和催化活性都是至关重要的。并且发现了其相互作用蛋白BLMH。BLMH通过与NUDT12二聚体形成异源十二聚体的形式控制NUDT12的定位从而参与到NUDT12的功能当中。本研究同时解析了人源NUDT12的分子结构,对解析NUDT12的去帽功能进行了进一步的详尽阐述。最后,本研究通过基因敲除技术,构建了NUDT12敲除小鼠模型。RNA测序表明,在NUDT12敲除小鼠的肝脏中,per1, per2, per3等生物钟相关基因上调,提示NUDT12对NAD-RNA的去帽作用对肝脏中的生物钟具有紧密的调节作用。
本研究通过分子生物学、生物化学、结构生物学以及基因敲除等技术从多方位的阐述了新的细胞质中的NAD-RNA去帽酶NUDT12,为了解NAD-RNA的生物合成与降解极其功能提供了更多的理论基础。
1. Sonenberg, N., Rupprecht, K.M., Hecht, S.M., and Shatkin, A.J. (1979). Eukaryotic mRNA cap binding protein: purification by affinity chromatography on sepharose-coupled m7GDP. Proc Natl Acad Sci U S A 76, 4345-4349.2. Chen, Y.G., Kowtoniuk, W.E., Agarwal, I., Shen, Y., and Liu, D.R. (2009). LC/MS analysis of cellular RNA reveals NAD-linked RNA. Nat Chem Biol 5, 879-881.3. Cahova, H., Winz, M.L., Hofer, K., Nubel, G., and Jaschke, A. (2015). NAD captureSeq indicates NAD as a bacterial cap for a subset of regulatory RNAs. Nature 519, 374-377.4. Walters, R.W., Matheny, T., Mizoue, L.S., Rao, B.S., Muhlrad, D., and Parker, R. (2017). Identification of NAD+ capped mRNAs in Saccharomyces cerevisiae. Proc Natl Acad Sci U S A 114, 480-485.5. Jiao, X., Doamekpor, S.K., Bird, J.G., Nickels, B.E., Tong, L., Hart, R.P., and Kiledjian, M. (2017). 5' End Nicotinamide Adenine Dinucleotide Cap in Human Cells Promotes RNA Decay through DXO-Mediated deNADding. Cell 168, 1015-1027 e1010.6. Zhang, H., Zhong, H., Zhang, S., Shao, X., Ni, M., Cai, Z., Chen, X., and Xia, Y. (2019). NAD tagSeq reveals that NAD(+)-capped RNAs are mostly produced from a large number of protein-coding genes in Arabidopsis. Proc Natl Acad Sci U S A 116, 12072-12077.7. Bird, J.G., Basu, U., Kuster, D., Ramachandran, A., Grudzien-Nogalska, E., Towheed, A., Wallace, D.C., Kiledjian, M., Temiakov, D., Patel, S.S., et al. (2018). Highly efficient 5' capping of mitochondrial RNA with NAD(+) and NADH by yeast and human mitochondrial RNA polymerase. Elife 7.8. McLennan, A.G. (2006). The Nudix hydrolase superfamily. Cell Mol Life Sci 63, 123-143.