传统制药工业生产过程工艺复杂
、环保压力大,且部分反应需要苛刻的条件。发展绿色制药技术,是打破环境污染这一限制制药行业发展瓶颈的关键[1]。近年来,随着酶制剂的规模化生产日益扩大,其在化学品生产中的应用也得到了快速发展。利用相应的酶,可以将化学途径中的多步反应通过一步酶促反应来实现,且具有高选择性、高效率、条件温和等优势。也正是因为这些优势,酶催化技术已经在部分药物中间体和原料药等的生产过程成功实现了产业化[2]。
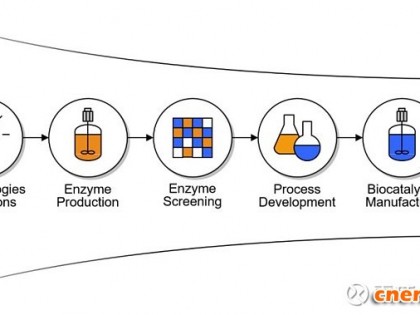
博腾秉承“客户第一,追求卓越”的核心价值观,为满足客户日益增长的希望用酶催化来更加绿色、高效、经济地生产药物中间体的需求,于2018年在上海研发中心搭建了包括酶催化技术团队在内的技术平台,并于2018年4月与世界领先的酶催化解决方案公司Codexis签订了战略合作协议。目前,博腾酶催化团队共有核心成员6人,将着力打造完善的技术能力(图1),为客户提供从路线创新、可行性研究、酶开发、制备、筛选、酶催化工艺开发和酶催化产品产业化放大等“一站式”CDMO酶催化解决方案。
图1 博腾酶催化技术能力
候选酶的高通量筛选
目前,博腾的酶库包括Codexis的酶,其他供应商来源的商品化酶以及具有博腾自主知识产权的酶。通过战略合作协议,Codexis向博腾开放了其包含1000余种工具酶的酶库使用权,博腾可以利用这些酶开展筛选、工艺优化等工作。除此之外,博腾也从其他的酶供应商处获得了商业化的酶来丰富自己的酶库资源。更重要的,博腾正积极建立自己独有知识产权的酶库,并已初具规模。博腾可以根据客户需要,从以上酶库中选择相应的酶进行筛选。
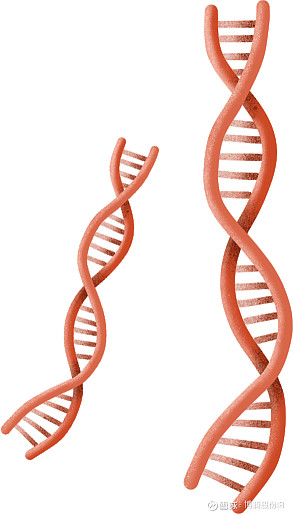
图2 高通量筛选流程示意图
进行酶促反应时,反应的结果可以通过检测底物的减少或产物的增加来判断。而这些检测通常需要专业的分析仪器,虽然可以精确地对结果做出判读,但往往时间较长,且需要繁琐的预处理操作,效率较低。有一些酶促反应,如氧化还原反应,通常需要如NAD(P)+等辅酶作为电子供体或受体,可以利用这些辅酶的性质(如特征性波长处吸光值的变化或颜色变化等)来初步判断反应结果。这些反应可以在多孔板(如96孔板)中用较小的体积(如50-200微升)进行反应。当颜色反应等初步结果显示获得了有催化活力的结果之后,将会通过液相或气相色谱等分析手段进一步鉴定产物,并计算催化效率(筛选流程图见图2)。目前,博腾已具备高通量筛选能力,可以在获得酶之后一周的时间内完成初筛工作。随着更多自动化设备到位,博腾的高通量筛选能力将更加强大。
酶的制备和化学品的酶催化制备
经过分析方法鉴定确认后的酶,将被用于进一步的酶催化工艺开发及优化,以提高酶的催化效率,降低成本。实验室规模酶制备(摇瓶+摇床)通常可以用于制备毫克或克级的酶制剂,足够用于实验室小体积的酶促反应,用以筛选一些酶促反应初步反应条件等;进一步,可以通过发酵罐(如1升,5升,10升等)来实现实验室规模的发酵,可获得10克级的酶制剂,可以用于酶促反应条件的实验室规模的放大条件优化,并用于制作展示用的小样化学品。同时,也可摸索并优化发酵条件,积累的大量发酵控制参数,为后期的中试放大(20升、300升等)以及最后的工业化生产放大(吨级)摸索条件。工业化的生产规模最大可达吨级,可用于制备公斤级的酶制剂;这些大量的酶制剂将被用于工厂规模的酶催化反应,大量制备化学品(示意图见图3)。
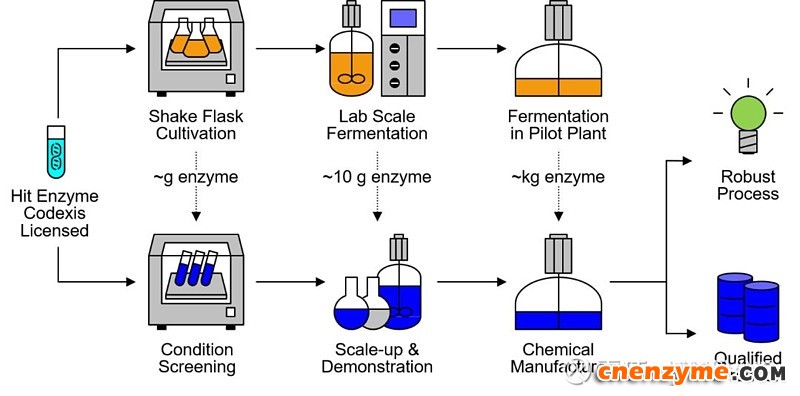
图3 酶的制备和化学品的酶催化制备
博腾完全按照项目的不同阶段对酶供应的不同需求,搭建了弹性高效的酶制备生产设备。目前,博腾已具备完善的实验室规模的发酵能力,中试发酵能力也初具雏形。预计2019年底,博腾将具备300升的发酵能力。届时,从只需少量酶量的(毫克或克级)实验室筛选(一周内完成),到实验规模(10克级)的发酵和催化工艺优化,再到工业级(公斤级)酶催化生产(周期视需求而定),博腾都将有能力提供灵活的解决方案,以满足客户不同规模的酶催化需求。
自有知识产权酶库的建立
及酶的定向进化
工业上使用的酶通常来源于微生物。在千百万年的进化过程中,微生物为了在自然环境中的各种选择压力下存活,物竞天择,进化出了可以在温和条件下实现各种满足代谢需求的酶。而酶的3D结构也让酶促反应有高度的底物特异性和空间选择性,不同来源的酶,对于相同底物的催化效率也可能有很大的不同。
最初的酶库,就是将文献中公开报道的一些在工业上有应用潜力的酶,如氨基转移酶、氧化还原酶、脂肪酶等等,用分子生物学的手段,将其在另外的宿主体内表达,如大肠杆菌、酵母等,可以大量获得目的蛋白。从这些天然的酶中,针对客户的需求开始筛选。天然酶可能一开始无法满足客户对高活力、高选择性、高ee值等指标的要求,所以在获得了可以初始的候选酶之后,有时需要借助生物信息学、结构生物学和分子生物学等手段对候选酶进行定向进化,以期获得具有更高的催化效率,更高的ee值,更高的稳定性等满足客户需求的酶。
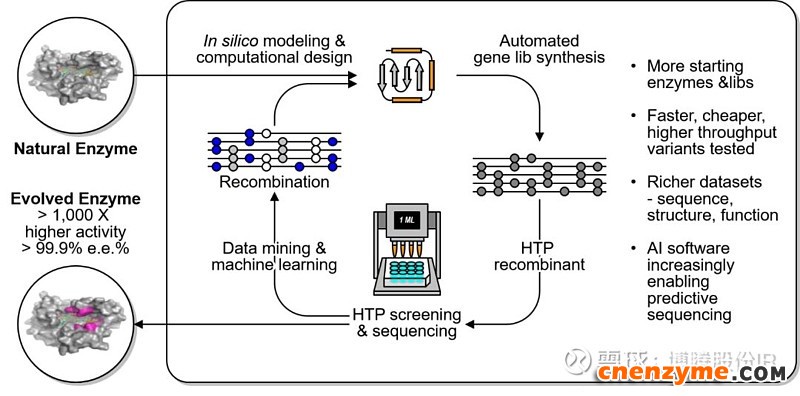
图4 计算机辅助的蛋白质定向进化
所谓酶的定向进化,即在实验室里通过随机突变和重组,人为制造大量的突变,按照特定的需要和目的给予选择压力,筛选出具有期望特征的蛋白质,实现分子水平的模拟进化。这个方法模拟了自然条件下的进化过程,使在自然条件下千百万年才可以完成的进化在实验室里极短的时间内完成。这是目前改善蛋白质性能最有效的方法[3]。
定向进化的方法,又可分为随机进化、半理性进化和理性进化。最初的定向进化,由于缺少酶的结晶结构的足够信息,所以通常用随机进化的方法,比如易错PCR,DNA shuffling等等[4-6],但存在文库庞大,阳性率不高等缺点。随着蛋白质结晶技术和计算机技术的发展,人们对蛋白质三维结构的认识更加深刻,于是就有了计算机辅助的半理性设计[7]和理性设计[8-11],可以对酶的活性位点区域进行有针对性的设计改造,大大减少无效克隆的数量,提高命中率。但无论半理性设计还是理性设计,都需要通过实验来验证,而实验反馈的信息又可以更好地辅助完善理性设计。近些年又出现了很多基于半理性设计的定向进化策略(如CAST/ISM)[12],以及发展迅速的人工智能技术(机器学习),也将有力助推蛋白质的定向进化工作。图4简单展示了计算机辅助的(半)理性设计的思路和流程。
目前博腾已经具备了完善的分子生物学实验能力,并在快速扩大自有知识产权的酶库;而酶进化的技术平台也日趋完善。


上海研发中心酶催化实验室
博腾酶催化实例
制药行业对酶催化技术的需求日趋旺盛,博腾也在积极响应市场需求,大力发展酶催化技术能力,并取得了长足的进步。
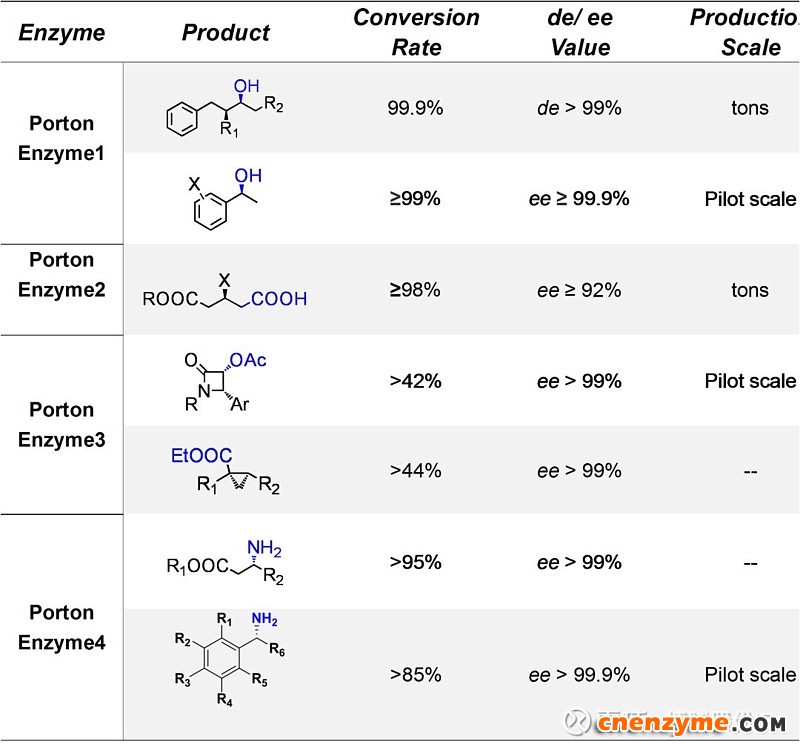
表1 博腾成功的案例
表1汇总了博腾成功完成的酶催化项目案例,赢得了客户的一致赞誉。博腾酶催化团队将持续加强自身能力建设,打造包括酶的挖掘、筛选、进化、工艺优化、酶制剂生产、酶催化化学品生产在内的完整技术能力,力争为客户提供“货架式”和“一站式”CDMO酶催化服务(图5)。

图5 博腾 “货架式”“一站式”CDMO酶催化服务
参考文献
[1] Seldon R A. The E factor:fifteen years on[J]. Green Chemistry,2007,9(12):1273-1283.
[2] Woodley J M. New opportunities for biocatalysis:making pharmaceutical processes greener[J]. Trends in Biotechnology,2008,26(6):321-327.
[3] Sheldon RA, Pereira PC. Biocatalysis engineering: the big picture. Chem Soc Rev, 2017, 46(10): 2678–2691.
[4] Leung DW, Chen E, Goeddel DV. A method for random mutagenesis of a defined DNA segment using a modified polymerase chain reaction. Technique, 1989, 1: 11–15.
[5] Hawkins RE, Russell SJ, Winter G. Selection of phage antibodies by binding affinity: mimicking affinity maturation. J Mol Biol, 1992, 226(3): 889–896.
[6] Ruff AJ, Dennig A, Schwaneberg U. To get what we aim for——Progress in diversity generation methods. FEBS J, 2013, 280(13): 2961–2978.
[7] Cheng F, Zhu LL, Schwaneberg U. Directed evolution 2.0: improving and deciphering enzyme properties. Chem Commun, 2015, 51(48):9760–9772.
[8] Ebert MC, Pelletier JN. Computational tools for enzyme improvement: why everyone can- and should-use them. Curr Opin Chem Biol, 2017, 37: 89–96.
[9] Huang PS, Boyken SE, Baker D. The coming of age of de novo protein design. Nature, 2016, 537(7620): 320–327.
[10] Bornscheuer UT, Huisman GW, Kazlauskas RJ, et al. Engineering the third wave of Biocatalysis. Nature, 2012, 485(7397): 185–194.
[11] Privett HK, Kiss G, Lee TM, et al. Iterative approach to computational enzyme design. Proc Natl Acad Sci USA, 2012, 109(10): 3790–3795.
[12] Reetz MT, Bocola M, Carballeira JD, et al. Expanding the range of substrate acceptance of enzymes: combinatorial active-site saturation test. Angew Chem Int Ed, 2005, 44(27): 4192–4196.