摘译自:https://www.nature.com/ncomms/2015/151124/ncomms10005/full/ncomms10005.html
构建生物分子合成途径首先要有高效的酶催化剂。2015年11月24号的Nature Communication文章Integrative genomic mining for enzyme function to enable engineering of a non-natural biosynthetic pathway提供了一种新的生物信息学和分子模拟相结合的功能酶基因组探矿方法。该方法该方法通用、便捷,筛选的酮酸脱羧酶GEO175与半理性设计的酶变体KIVD一样,均能将工程菌合成短链醇转向长链醇的合成。
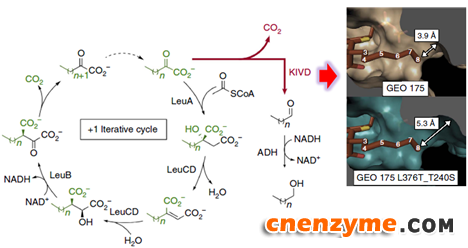
乳酸乳球菌的2-酮异戊酸脱羧酶(KIVD,2-ketoisovalerate decarboxylase)可催化酮异戊酸生成异丁醇(C4),KIVD与异丙基苹果酸合酶 (LeuA),异丙基苹果酸异构酶复合体(LeuC, D),异丙基苹果酸脱氢酶(LeuB)构建的循环+1途径可合成甲基丁醇(C5)和甲基戊醇(C6)等长链醇(https://www.pnas.org/content/early/2008/12/08/0807157106 )。
而进一步合成更高级的醇(C8),需要寻找KIVD同源酶或对KIVD进行理性或半理性重设计,以获得催化长链酮酸脱羧的酮酸脱羧酶(ketoacid decarboxylases),构建非天然的长链醇合成途径。
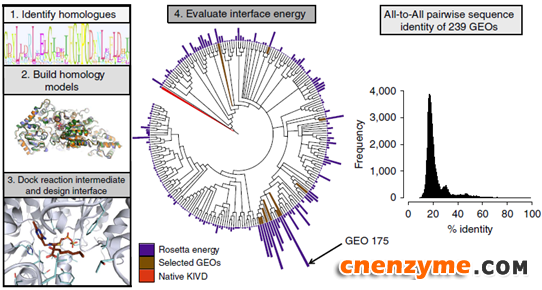
图2:计算机介导基因组功能酶探矿的流程
基因组功能酶探矿新方法如图2示:
1)序列比对得到2082个KIVD的同源酶(genomic enzyme orthologues: GEOs),并剔除彼此同源性大于90%及真核来源的GEOs;
2)用 Rosetta Comparative Modeling同源模建,用 TMalign算法进一步筛选共获得239个GEOs,这239个序列间同源性约20%;
3)239个GEOs与C8底物进行分子对接(docking),根据酶与底物间的表面能(interface energy)大小创建进化树,进化树显示GEO175表面能最低,即最有可能催化C8底物;
3)根据表面能及同源性,选10个GEOs进行动力学表征,6个获得可溶蛋白,3个对酮酸(C3, C5, C8, KIVD天然底物中的至少一种)有可检测到的活性。其中GEO175对C8/C5的特异性比KIVD提高100倍。
基于结构及突变分析,研究者认为GEO175相比KIVD,其结合口袋与C6-C8底物的强疏水作用决定了其底物特异性。
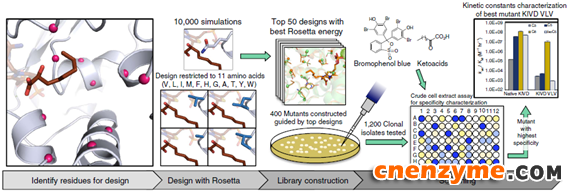
图3:对KIVD半理性设计筛选底物特异性改变突变体的流程
传统半理性设计方法如图3示:利用Rosetta Design对KIVD的活性位点重设计,选择10个氨基酸位点分别突变为11种疏水氨基酸,从10000个可能的设计中选出50个能值低的模型,构建约400个突变体库,筛选1200个转化子进行活性及特异性测试。提高底物特异性的突变位点组合得到突变体G402V/M538L/F542(KIVD_ VLV),KIVD_VLV对C8/C5的特异性比KIVD提高600倍,而催化效率基本没变。
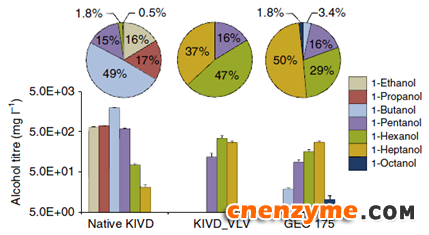
图4 携带不同酮酸脱羧酶工程菌的醇生成情况
最后,KIVD_VLV及GEO175用于大肠杆菌工程菌,测试长链醇生成情况(图4)。KIVD的产物中82%是C2-C4的短链醇。而KIVD_VLV完全转向合成C5-C7长链醇,共728 mg/L。GEO175产物有96%为C5-C8长链醇,其中C7醇274 mg/L,占50%。而且,只有GEO175合成C8醇。